|
Nghiên cứu ứng dụng phương pháp tổng hợp DNA sản phẩm đa mồi trong chẩn đoán phân tử xác định và phân biệt ký sinh trùng
Phương pháp hình thái học sử dụng kiểu hình trong phân loại là thông dụng và có những thành tựu to lớn trong chẩn đoán xác định và phân biệt ký sinh trùng, nhưng khó khăn gặp phải là khó phân biệt loài và dưới loài hay các dạng phát triển (trứng, metacercaria…) một cách chính xác. Trong hai thập kỹ qua, công nghệ ứng dụng kỹ thuật mới về sinh học phân tử đã có những định hướng phát triển vượt bậc, trước hết là kỹ thuật nhân bản DNA, đó là PCR (polymerase chain reaction) và kỹ thuật LAMP (loop-mediated isothermal amplification). PCR đa mồi (multiplex-PCR) và LAMP đã là những công cụ hữu hiệu được ứng dụng rộng rãi trong y-sinh học, trong đó có chẩn đoán phân tử ký sinh trùng. Cả hai phương pháp PCR/RT-PCR (multiplex) và LAMP/RT-LAMP đều sử dụng: i) Cùng lúc nhiều mồi (từ 2 mồi trở lên); ii) Acid nucleic cung cấp vùng gen đích làm khuôn là DNA hay RNA; iii) Deoxy-nucleotide triphosphate (dNTP) là các dẫn chất làm nguyên liệu kiến tạo; iv) Tổng hợp chuỗi nucleotide được xúc tác bởi một loại enzyme DNA polymerase; v) Phản ứng hoạt động được nhờ các thành phần làm phụ trợ trước hết là ion Mg++; vi) Xác nhận kết quả bằng cách đánh giá sự hiển thị của sản phẩm DNA thích hợp. Hai phương pháp này có những lợi thế khi sử dụng, ở những điều kiện nhất định. Trong bài tổng quan này, chúng tôi xin giới thiệu tầm quan trọng của PCR và LAMP, nguyên tắc hoạt động của multiplex-PCR và LAMP và những ứng dụng trong chẩn đoán phân tử bằng phương pháp tổng hợp DNA sản phẩm đa mồi để xác định và phân biệt ký sinh trùng. Bài tổng quan phương pháp này được hình thành trên cơ sở chủ yếu dựa vào tài liệu/dữ liệu của những nghiên cứu trên thế giới với mong muốn được ứng dụng rộng rãi hơn nữa trong lĩnh vực nghiên cứu và chẩn đoán ký sinh trùng tại Việt Nam. Từ khóa: PCR, mồi, multiplex-PCR, LAMP, DNA mạch thẳng/mạch vòm, ký sinh trùng. ĐẶT VẤN ĐỀ Chẩn đoán, giám định và phân loại nhiều loại sinh vật, trong đó có ký sinh trùng cho đến gần đây chủ yếu vẫn dựa vào xác định đặc tính hình thái học là chính. Mặc dầu hình thái học (kiểu hình) trong phân loại là rất thông dụng và đã có những thành tựu hết sức to lớn, nhưng trong nhiều trường hợp khó khăn gặp phải là khó phân biệt loài và dưới loài một cách chính xác (McCarthya, Moore, 2000). Do tiêu tốn thời gian và khó tìm kiếm đúng môi trường thích hợp nuôi cấy cho một số loại vi khuẩn/virus, hay vật chủ cho ký sinh trùng, bệnh phẩm đòi hỏi phải hoàn chỉnh hoặc phải ở trạng thái sống, nên các phương pháp chẩn đoán dựa trên nuôi cấy và hình thái học có rất nhiều hạn chế (Gilbert, 2002; Parida, 2008; Mori và Notomi, 2009). Các phương pháp chẩn đoán truyền thống áp dụng trong lâm sàng và dịch tễ học như xét nghiệm tìm trứng/ấu trùng hay các sản phẩm của ký sinh trùng hoặc các kỹ thuật miễn dịch học đều có những sai số nhất định, tuỳ từng loài ký sinh trùng, đặc biệt khi các dạng ký sinh trùng cùng tồn tại ở một nơi như đường ruột ở hệ tiêu hóa, chất thải ở hệ hô hấp hay máu của hệ tuần hoàn. Phương pháp sinh học phân tử nhân bản DNA và phân tích đặc tính sản phẩm thu được, đặc biệt là phân tích biến đổi gen/hệ gen được ứng dụng trong chẩn đoán phân loại và lập phả hệ sinh vật, đã bước đầu chứng minh cho kết quả chính xác hơn nhiều so với phương pháp truyền thống (Elnifro et al., 2000; De Lellis et al., 2008; Mori và Notomi, 2009). Các số liệu chính xác của các phương pháp sinh học phân tử đã cho phép ứng dụng những hướng mới và rất có thể sẽ làm thay đổi một phần hệ thống phân loại và chẩn đoán phân biệt loài hiện có (Galluzzi et al., 2007; Littlewood, 2008). Trong hai thập kỹ qua, công nghệ ứng dụng kỹ thuật mới đã có những định hướng phát triển vượt bậc. Một trong những thành tựu lớn của nhân loại là sự phát triển phương pháp nhân bản DNA bằng kỹ thuật đa mồi (multiplexing methodology), đặc biệt những phương pháp nhân bản DNA sử dụng nhiều mồi cùng một lúc phát hiện đa khuôn đó là kỹ thuật multiplex-PCR (multiplex polymerase chain reaction) tạo sản phẩm DNA mạch thẳng (Powledge, 2004; Ishmael, Stellato, 2008) và kỹ thuật LAMP (loop-mediated isothermal amplification) đa mồi nhân bản DNA phát hiện đơn khuôn, tạo sản phẩm DNA cấu trúc vòm (Gill, Ghaemi, 2008; Parida et al., 2008; Mori, Notomi, 2009). Nếu sử dụng RNA làm khuôn, sau khi chuyển đổi sang DNA bằng phản ứng sao chép ngược (reverse transcription), cả PCR và LAMP cũng đều có thể thực hiện quá trình tổng hợp chuỗi nucleotide cho sản phẩm DNA. Do vậy, bên cạnh PCR sử dụng khuôn DNA, còn có RT-PCR sử dụng khuôn RNA; và bên cạnh LAMP sử dụng khuôn DNA, còn có RT-LAMP sử dụng khuôn RNA (Goto et al., 2007; Parida et al., 2008). Cả hai phương pháp PCR/RT-PCR và LAMP/RT-LAMP đều sử dụng cùng lúc nhiều mồi (từ 2 mồi trở lên); sử dụng DNA hay RNA làm khuôn cung cấp vùng gen đích; sử dụng các dẫn chất deoxy-nucleotide triphosphate (dNTP) làm nguyên liệu kiến tạo; sử dụng một loại DNA polymerase làm enzyme để xúc tác tổng hợp chuỗi nucleotide; sử dụng các thành phần làm phụ trợ cho phản ứng hoạt động trong đó có i-on Mg++ có vai trò quan trọng; và xác nhận kết quả bằng cách đánh giá sự hiển thị của sản phẩm DNA trong điều kiện thích hợp (Lê Thanh Hòa, Trương Nam Hải, 2010). CHẨN ĐOÁN PHÂN TỬ KÝ SINH TRÙNG BẰNG KỸ THUẬT PCR ĐA MỒI Giới thiệu phương pháp PCR Trong 20 năm nay, nghiên cứu sinh học phân tử được đẩy mạnh nhờ sự phát minh PCR và PCR được coi là kỹ thuật đa dụng và hiệu quả nhất (Powledge, 2004; Ishmael, Stellato, 2008). PCR là phản ứng phụ thuộc chu kỳ nhiệt độ qua nhiều giai đoạn khác nhau, có enzyme DNA polymerase chịu nhiệt xúc tác, sử dụng nhiều nhất là Taq polymearase (phân lập từ vi khuẩn chịu nhiệt Thermus aquaticus), trong môi trường có nồng độ i-on Magiê (Mg++) thích hợp, với các thành phần dẫn chất deoxy-nucleotide triphosphate (dNTP) đầy đủ làm nguyên liệu, có hai đoạn ngắn nucleotide làm mồi (primer) và sản phẩm tạo ra là DNA mạch thẳng (linear DNA). Chỉ cần một lượng nhỏ DNA (tinh khiết hoặc hỗn hợp) làm khuôn, phản ứng PCR sẽ tiến hành tổng hợp các đoạn DNA tương ứng trên khuôn đó, cho sản phẩm là các đoạn DNA trong giới hạn giữa các mồi. Sự tổng hợp DNA tăng theo cấp số 2n, sau 25 - 40 chu kỳ, số lượng DNA đạt được là hàng tỷ bản sao. Thời gian thực hiện PCR là vài giờ, với chương trình tự động hoá, nên không tốn công sức (Ishmael, Stellato, 2008). Hiện nay, đã có máy phân tích trình tự nucleotide tự động (automated sequencer), nên sản phẩm PCR sau khi được làm sạch, sẽ được phân tích ngay rất thuận lợi cho nghiên cứu. Phương pháp PCR không đòi hỏi DNA khuôn với lượng lớn, chỉ cần một lượng nhỏ mẫu ký sinh trùng (mg) thậm chí chỉ cần một ít trứng giun sán là đã có thể tách chiết DNA đủ để làm khuôn mà không cần phải nuôi cấy (khoảng 0,1 -100 ng ADN làm khuôn là đủ), PCR được ứng dụng cho giám định, chẩn đoán, phân loại, di truyền quần thể, phả hệ và tiến hoá sinh vật, trong đó có ký sinh trùng (Chandler và Colitz, 2006; Littlewood, 2008) và trở thành một công cụ không thể thiếu được trong công tác giám định, phát hiên sinh vật (kể cả ký sinh trùng) gây bệnh bằng kỹ thuật cao. Dễ làm, có tính nhạy và đặc hiệu rất cao, đồng thời chỉ cần một lượng rất ít khuôn DNA của đối tượng sinh vật bất kể giai đoạn sinh trưởng nào, PCR đã có thể cho sản phẩm với độ chính xác cao về loại sinh vật cần nghiên cứu. Với lợi thế như vậy, PCR đã thực sự được ứng dụng rộng rãi trong mọi lĩnh vực, trước hết là chẩn đoán phân tử xác định và phân biệt các loài gây bệnh (Ishmael và Stellato, 2008). Có thể phân biệt có 2 loại PCR chính theo qui mô kỹ thuật, đó là PCR thông thường và PCR đặc biệt. PCR thông thường (conventional PCR) là sử dụng một (hay vài) cặp mồi đặc hiệu, cho sản phẩm DNA thu được có độ dài vài trăm đến vài nghìn nucleotide (Ishmael và Stellato, 2008); còn PCR đặc biệt (special PCR) là các dạng PCR sử dụng các thành phần đặc biệt với hai hay nhiều mồi, thu sản phẩm có kích thước dài ngắn khác nhau (Powledge, 2004). PCR đặc biệt trước hết phải kể đến: i) PCR dài (long PCR): cho sản phẩm vài chục nghìn nucleotide, thành phần tham gia đòi hỏi tinh khiết, chọn lọc, có tính năng cao, thường được sử dụng trong nghiên cứu giải mã một vùng gen lớn hoặc toàn bộ hệ gen (Le et al., 2002; De Lellis et al., 2008); ii) PCR lồng (nested PCR): có PCR bán lồng (semi-nested PCR) sử dụng 3 mồi; PCR lồng hoàn toàn (fully nested PCR) sử dụng 2 hay nhiều cặp mồi trong một phản ứng và PCR lồng đảm bảo tạo sản phẩm một cách chắc chắn vì DNA sinh ra ở chu kỳ trước của PCR làm nguồn khuôn tiếp tục cho các chu kỳ sau (Coiras et al., 2004); iii) PCR đơn và kép: PCR đơn (uniplex-PCR) sử dụng một cặp mồi, tìm kiếm sản phẩm trên một nguồn khuôn từ một loại (vi) sinh vật; PCR kép (duplex-PCR) sử dụng 2 cặp mồi, tìm kiếm sản phẩm trên 2 nguồn khuôn từ 2 loại (vi) sinh vật (Deng et al., 2000); iv) PCR đa mồi: PCR đa mồi (multiplex-PCR) sử dụng nhiều cặp mồi, tìm kiếm sản phẩm trên nhiều vùng đích khác nhau của cùng một khuôn hoặc của nhiều nguồn khuôn khác nhau, do đó, cùng lúc chẩn đoán, phát hiện nhiều gen của nhiều loại (vi)sinh vật và ký sinh trùng khác nhau. Tuỳ theo số lượng gen đích cần chẩn đoán mà multiplex-PCR được chia làm các loại là duplex-PCR: phát hiện 2 gen; triplex-PCR: phát hiện 3 gen; quadruplex-PCR: phát hiện 4 gen; pentaplex-PCR: phát hiện 5 gen; hexaplex-PCR: phát hiện 6 gen v.v... Nói chung, phát hiện 2 gen trở lên đều gọi là PCR đa gen và loại hình multiplex-PCR này chính là phương pháp nền của việc xây dựng kit chẩn đoán phân tử đáp ứng mục tiêu của đề tài đặt ra (Edwards, Gibbs, 1994; Kaderali, 2007); Ngoài ra còn nhiều loại PCR khác như sử dụng mồi không đặc hiệu hay PCR xác suất (random PCR), PCR cực đại (mega PCR), PCR tăng/giảm nhiệt độ, PCR định lượng (real-time PCR), PCR mỏ neo (anchor-PCR), PCR đảo ngược (inverse-PCR), đều được sử dụng trong nghiên cứu. PCR định lượng (real-time PCR, ký hiệu rtPCR) cũng có thể phát triển thành rtPCR đa mồi (real-time multiplex-PCR) để chẩn đoán phân biệt và định lượng RNA thông tin từ nguồn khuôn nucleic acid có cấu trúc là RNA, tuy nhiên giá thành cao chỉ chấp nhận trong thực tế với những trường hợp đặc biệt (Kubista et al., 2006; Haque et al., 2007). Đối với nghiên cứu phân tử ký sinh trùng chẩn đoán xác định và phân biệt với mục đích giám định phân loại, chúng ta thường sử dụng PCR thông thường, PCR lồng, PCR đa mồi và real-time PCR (Kubista et al., 2006). Đặc điểm của multiplex-PCR (PCR đa mồi) Mỗi một lần làm phản ứng chẩn đoán sử dụng PCR đơn gen (uniplex-PCR) để phát hiện thì tiêu tốn thời gian, nguồn khuôn, công sức, tiền của và chưa được kinh tế, hơn nữa làm chậm tiến độ chẩn đoán. Do vậy, gần đây người ta đã phát triển ứng dụng PCR đa mồi phát hiện đa gen (multiplex-PCR) cùng lúc phát hiện sự có mặt của nhiều sinh vật trong một nguồn bệnh phẩm, cho dù đó là khuôn DNA tổng số gồm nhiều loại khác nhau, lấy từ các cá thể khác nhau cần chẩn đoán (Elnifro et al., 2000). PCR đa mồi (multiplex-PCR) được hiểu theo nghĩa đơn giản nhất, đó là nhiều thành phần mồi sử dụng (đa mồi), nhiều số lượng vùng gen đích cần phát hiện (đa gen), nhiều số lượng loài cần phát hiện (đơn hoặc đa loài) và nhiều số lượng sản phẩm DNA mạch thẳng được tạo ra (có từ 2 trở lên) (Hình 1). | 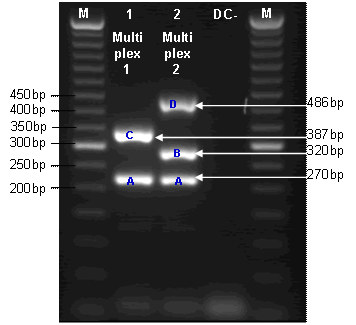
| Hình 1. Kiểm tra sản phẩm multiplex-PCR (đa mồi, phát hiện đa gen từ nguồn khuôn đa loài) trên thạch agarose 1%, giả sử ở đây sử dụng 4 cặp mồi, phát hiện 4 loài khác nhau, ký hiệu A, B, C, D. Ghi chú: M: marker, chỉ thị phân tử, ở đây sử dụng thang DNA cách nhau 50 bp; DC-: đối chứng âm, không có khuôn; 1: Multiplex-1, sản phẩm thu được từ hỗn hợp khuôn bệnh phẩm 1, đã phát hiện chỉ có sản phẩm PCR của 2 loài là 270 bp của loài A và 387 bp của loài C, không có loài B và D; 2: Multiplex-2, sản phẩm thu được từ hỗn hợp khuôn bệnh phẩm 2, đã phát hiện có sản phẩm PCR của 3 loài là 270 bp của loài A, 320 bp của loài B và 486 bp của loài D, không có loài C được phát hiện. Như vậy, loài A được phát hiện có trong cả hai bệnh phẩm. |
Về nguyên tắc, trong PCR đa mồi/đagen, cùng lúc các chuỗi gen của cùng một đối tượng gây bệnh hay của các đối tượng khác nhau đều có thể nhân lên được, bằng cách sử dụng nhiều cặp mồi đặc hiệu trong cùng một phản ứng. Mỗi một cặp mồi đặc hiệu sẽ tìm kiếm chính xác vùng gen đích và nhân vùng gen đó để cho sản phẩm. Nếu thiết kế cặp mồi đặc hiệu riêng cho mỗi vùng gen đích cần chẩn đoán (của cùng loài hay khác loài), nếu sản phẩm PCR thu được có kích thước chênh lệch nhau, thì khi sản phẩm PCR hiển thị trên agarose, rất dễ dàng đánh giá phân biệt. Kích thước của sản phẩm phải chênh lệch nhau ít nhất 50 bp trở lên mới đánh giá được khi điện di trên thạch (Deng et al., 2000; Kaderali, 2007) (Hình 1). Đại đa số trường hợp, multiplex-PCR được sử dụng phát hiện 3 - 4 gen đích của một loài hoặc các loài sinh vật; nhiều trường hợp đặc biệt, người ta sử dụng nhiều mồi hơn để phát hiện một lúc 6 nguồn khuôn (hexaplex-PCR) (Singh, 2003), hayxác định 7 loài khác nhau (Fernandez et al., 2003), thậm chí có nhiều khi phát hiện đến 14 loại virus khác nhau với độ nhạy và chính xác cao (Coiras et al., 2004). Nhờ những lợi thế đó, multiplex-PCR đã chứng tỏ là phương thức chẩn đoán phân tử tiết kiệm thời gian, công sức, nguồn khuôn từ bệnh phẩm, đa dụng, phát hiện đa bệnh, dễ làm, tiện ích (Edwards, Gibbs, 1994; Elnifro et al., 2000). Chính vì vậy, ngày nay, chẩn đoán phân tử dựa trên cơ sở chuỗi nucleotide (DNA và RNA) bằng kỹ thuật PCR đa mồi (đối với khuôn là DNA) hoặc/và RT-PCR đa mồi (đối với khuôn là RNA), kể cả kỹ thuật định lượng nguồn gen (real-time PCR/RT-PCR) đã được ứng dụng để phân tích gen/hệ gen, phân tích đa hình, chẩn đoán và phân tích nhiễm sắc thể, chẩn đoán vi sinh vật gây bệnh, ký sinh trùng, nấm, vi khuẩn, độc tố, kháng thuốc, định lượng RNA, đánh giá chất lượng sản phẩm, xác định sinh vật chuyển gen và nhiều mục đích khác (Deng et al., 2000; Kubista et al., 2006; ono et al., 2007; De Lellis et al., 2008). Về loại hình multiplex-PCR, được sử dụng nhiều nhất là loại phản ứng multiplex-PCR sử dụng nhiều cặp mồi, mà mỗi cặp phát hiện đặc hiệu mỗi một gen đích tương ứng, với độ nhạy cao, nhưng nhờ có sản phẩm PCR thu được có kích thước khác nhau, cho nên, khi điện di trên thạch agarose các sản phẩm phân biệt dễ dàng ứng với mỗi loài đích cần chẩn đoán (Deng et al., 2000). Nghiên cứu phát triển multipex-PCR theo hướng này, đòi hỏi thiết kế mồi và thử/kiểm nghiệm phản ứng phải hết sức chuẩn xác (Kaderali et al., 2007), nhưng một khi đã có bộ sinh phẩm (kit), thì ứng dụng chẩn đoán xét nghiệm rất có hiệu quả và kinh tế (Parida et al., 2008). PCR đa mồi (multiplex-PCR) trong chẩn đoán phân tử xác định và phân biệt ký sinh trùng Đối với ký sinh trùng, cho đến nay đã có hàng chục phương pháp và kit chẩn đoán multiplex-PCR sử dụng phát hiện nhiều loài gây bệnh. Khi ký sinh trùng cùng tồn tại ở một vị trí, một vùng trong cơ quan của cơ thể (ví dụ: cùng ở trong máu, cùng ở trong đường ruột/khoang ruột, cùng ở trong phổi/dịch phổi, hoặc metacercariae cùng ở trong một vật chủ trung gian), thì việc cùng lúc phát hiện và phân biệt được nhiều loài gây bệnh mang lại tính kinh tế và lợi ích cao hơn nhiều so với thực hiện đơn lẻ (Edwards, Gibbs, 1994; De Lellis et al., 2008). Multiplex-PCR đã được xây dựng để ứng dụng chẩn đoán phân biệt các loài sán lá/sán dây (platyhelminth), bao gồm sán lá gan nhỏ Clonorchis sinensis and Opisthorchis viverrini (Le et al., 2006); sán lá gan lớn F. hepatica từ ốc vật chủ trung gian (Magalhães et al., 2004; 2008); sán lá phổi Paragonimus spp (Sugiyama et al., 2006; Le et al., 2006); sán dây Taenia saginata, Taenia solium, Taenia saginata asiatica và ấu trùng sán lợn (González et al., 2000; 2004; Yamasaki et al., 2004; Trachsel et al., 2007); sán chó Echinococcus multilocularis (Davidson et al., 2009). Multiplex-PCR cũng được sử dụng phát hiện và phân biệt các loại giun tròn (nematode) trong đó có Trichinella và nematode đường ruột (Zarlenga et al., 1999; 2001a,b); phân biệt Ancylostoma duodenale, Necator americanus, Oesophagostomum bifurcum (Verweij et al., 2007) hoặc các loài Oesophagostomum spp ở lợn (Lin et al., 2008); phát hiện và phân biệt Brugia malayi và Wuchereria bancrofti với nhau (Mishra et al., 2007) và với ký sinh trùng sốt rét (Rao et al., 2009); Multiplex-PCR tỏ ra sử dụng rất hữu hiệu chẩn đoán phân biệt nhiều nhóm ký sinh trùng đơn bào (protozoa), chủ yếu tập trung vào nhóm ký sinh trùng đơn bào đường ruột như Cryptosporidium và Giardia (Patel et al., 1999; Gile et al., 2002; Guy et al., 2003); Cyclospora và Eimeria (Orlandi et al., 2003; Fernandez et al., 2003); đặc biệt là ký sinh trùng đơn bào máu, chủ yếu tập trung ký sinh trùng sốt rét Plasmodium spp, trước hết là phân biệt 4 loài gây bệnh, chủ yếu ở người, đó là Plasmodium falciparum, P. vivax, P. malariae, P. ovale, theo từng nhóm hoặc cả 4 loại từ máu (Padley et al., 2003; Patsoula et al., 2003; Rougemont et al., 2004; Veiga et al., 2006; Rao et al., 2009); hoặc từ vector là muỗi Anopheles (Lardeux et al., 2008). Một loại ký sinh trùng khác thường có trong máu là Leishmania spp cũng được chẩn đoán phân biệt bằng multiplex-PCR từ máu, dưới da hay từ nguồn truyền lây vector là loài muỗi cát Lutzomyia (Harris et al., 1999; Jorquera et al., 2005; Rodríguez-González et al., 2007; de Pita-Pereira et al., 2008) hayký sinh trùng đường máu lê dạng trùngBabesia caballi và Babesia equi ở động vật (Alhassan et al., 2005) hoặc các loài đơn bào máu khác cũng được phát hiện bằng multiplex-PCR trong đó có Toxoplasma gondii (Ajzenberg et al., 2005; Nowakowska et al., 2006). Multiplex-PCR còn được ứng dụng chẩn đoán lỵ amíp do khả năng phát hiện nhanh, chính xác phân biệt các loài gây bệnh với nhiều loài khác có hình thái tương tự, trước hết là Entamoeba histolytica và Entamoeba dispar từ phân người bệnh (Evangelopoulos et al., 2000; Haque et al., 2007; Khairnar, Parija, 2007). Về vùng gen đích chọn lựa sử dụng chẩn đoán, nhiều tác giả khai thác hệ gen nhân tế bào trong đó hướng tới đối tượng gen 18S rRNA phân biệt cầu trùng Eimeriavới Cyclospora(Orlandi et al., 2003), giám định và phân biệt các loài Cryptosporidium (Patel et al., 1999), phát hiện các loài ký sinh trùng sốt rét Plasmodium (Rougemont et al., 2004); hay đối tượng là gen pfcrt, pfmdr1, pfdhfr ở ký sinh trùng sốt rét (Veiga et al., 2006) và nhiều vùng gen hay vùng giao gen khác. Nhiều công trìnhkhai thác đích là hệ gen ty thể (mitochondrial genome), ví dụ chẩn đoán phân biệt sán dây Taenia spp (Trachsel et al., 2007; Yamasaki et al., 2004; 2006); sán lá gan nhỏ Clonorchis sinensis và Opisthorchis viverrini (Le et al., 2006); sán lá gan lớn Fasciola hepatica (Magalhães et al., 2004; 2008); phân biệt định loài động vật trong thức ăn thực phẩm (Ono et al., 2007). Hướng sử dụng hệ gen ty thể trong nghiên cứu chẩn đoán loài kể cả ứng dụng multiplex-PCR được coi là có cơ sở vững vàng trong điều kiện hiện nay. Do có kích thước nhỏ, tồn tại nhiều bản sao trong một tế bào (vài trăm/tế bào), cấu trúc là vòng DNA khép kín nên rất bền vững, cấu trúc đơn gen liên tục thuận lợi cho PCR hoạt động, một tế bào cho nguồn khuôn DNA gấp hàng trăm lần so với hệ gen nhân nên phản ứng PCR có độ nhạy cao hơn (Le et al., 2002; Hu, Gasser, 2006). Điều này có ý nghĩa khi sử dụng tế bào trứng giun sán làm nguồn khuôn cho multiplex-PCR, vì chỉ cần 1 tế bào trứng đã cho hàng trăm phân tử DNA hệ gen ty thể. Nhiều công trình đã thiết kế multiplex-PCR chỉ sử dụng trứng giun/sán cung cấp nguồn khuôn DNA để tận dụng tính đơn giản thu mẫu (chỉ cần phân người bệnh, đối với nhiều ký sinh trùng phủ tạng và đường ruột; hay đờm chất dịch hô hấp, đối với sán lá phổi) (Trachsel et al., 2007; Davidson et al., 2008). Gen/hệ gen ty thể của nhiều loài ký sinh trùng đã lần lượt được giải mã và đăng ký trong Ngân hàng gen thế giới (http://www.ncbi.nlm.nih.gov), tạo cơ sở dữ liệu để có thể truy cập ứng dụng trong việc thiết kế mồi đặc hiệu từng loài cho multiplex-PCR và so sánh phân biệt trong chẩn đoán, giám định và phân loại (Le et al., 2002; Hu, Gasser, 2006). Có hàng ngàn hệ gen ty thể đã được giải trình tự và phân tích hoàn toàn, hàng trăm hệ gen ty thể khác đang từng phần được giải quyết, bao gồm tất cả mọi loài quan trọng từ bậc thấp đến bậc cao, từ động vật đơn bào đến động vật đa bào, cung cấp một hệ thống dữ liệu quan trọng cho các quá trình nghiên cứu gen, protein, tiến hoá, lịch sử tiến hoá, di truyền quần thể, quan hệ về loài, giống và nhiều lĩnh vực nghiên cứu quan trọng khác, trong đó có sử dụng dữ liệu để thiết kế mồi cho multiplex PCR. Ngoài ra còn có thể sử dụng các gen đích của hệ gen nhân trong chiến lược thiết kế phương pháp PCR đa gen/đa mồi để chẩn đoán phân biệt. Nghiên cứu ứng dụng PCR đa mồi xác định và phân biệt ký sinh trùng tại Việt Nam Nghiên cứu về sinh học phân tử giám định thành phần loài và chẩn đoán phân biệt các loài giun/sán ở Việt Nam bằng kỹ thuật PCR sử dụng chỉ thị di truyền hệ gen ty thể đã được một số tác giả thực hiện có hiệu quả, đặc biệt trong 10 năm gần đây. Có hàng trăm công trình sử dụng PCR đa mồi trong chẩn đoán phân biệt ký sinh trùng được đăng tải phản ánh định hướng nghiên cứu ứng dụng kỹ thuật cao đối với lĩnh vực ký sinh trùng, mà chúng tôi không có ý định nêu hết trong bài tổng quan này, chỉ điểm qua một số kết quả do nhóm chúng tôi thực hiện được. Trước hết, đó là nghiên cứu xác định loài sán dây châu Á Taenia asiatica đầu tiên ở Việt Nam (Lê Thanh Hòa và cs, 2002; Nguyễn Văn Đề, Lê Thanh Hoà, 2006). Sau đó các loài sán khác lần lượt đã được thẩm định thành phần loài như: sán lá gan nhỏ C. sinensis, sán lá ruột Fasciolopsis buski, sán lá gan lớn Fasciola gigantica, sán lá phổiParagonimus heterotremus, P. vietnamensis, P. proliferus (Le và cs, 2006; Doanh và cs, 2007; 2008; 2009); sán dây T. saginata, T. solium ký sinh trên người, ấu trùng sán dây lợn trên lợn và trên người cũng đã được thẩm định bằng sinh học phân tử hệ gen ty thể (Lê Thanh Hoà và cs, 2002; Nguyễn Văn Đề, Lê Thanh Hoà, 2006) và lần lượt một số chuỗi gen đã đăng ký trong Ngân hàng Gen thế giới. Những kết quả bước đầu này đã góp phần giải quyết một số vấn đề trong xác định thành phần loài giun sán tại Việt Nam và đã làm cơ sở khoa học cho việc nghiên cứu kit multiplex PCR để chẩn đoán xác định bệnh giun sán. Đồng thời đó cũng là cơ sở dữ liệu quý báu cho khoa học trong nghiên cứu đa dạng sinh học và đóng góp vào hoạch định chiến lược phòng chống giun sán có hiệu quả ở Việt Nam. Cho đến nay, nghiên cứu sinh học phân tử trong chẩn đoán ký sinh trùng mới thực hiện ở góc độ đơn loài, kit PCR đơn gen, mà chưa có những định hướng tổng thể sàng lọc, xây dựng kit đa gen (multiplex-PCR) cho một nhóm cùng loài, hay cho một hỗn hợp các loài cùng tồn tại trên người ở những vị trí phân bố cần chẩn đoán (Lê Thanh Hoà, 2007). Tuỳ theo khu trú hỗn hợp của các loài ký sinh trùng gây bệnh, chúng tôi mong muốn chọn lọc phân nhóm loài đối tượng, hoặc/và phân vùng khu trú (ruột, lấy mẫu phân; phổi, lấy mẫu đờm, dịch tiết phổi), xây dựng kit song gen (duplex-PCR), đa gen (multiplex-PCR) để chẩn đoán sàng lọc và giám định và đó cũng chính là những định hướng xuyên suốt trong chiến lược xây dựng phương thức và kit chẩn đoán bằng sinh học phân tử ở nước ta. Do vậy, việc nghiên cứu chẩn đoán phân tử là hết sức bức thiết, đặc biệt là nghiên cứu kit PCR đa gen/đa mồi (multiplex-PCR) đối với ký sinh trùng mà chúng tôi đang xây dựng sẽ giúp cho việc chẩn đoán hoàn toàn chính xác và góp phần to lớn trong phòng chống bệnh có hiệu quả trong phạm vi cả nước (Lê Thanh Hòa, 2009). CHẨN ĐOÁN PHÂN BIỆT KÝ SINH TRÙNG BẰNG KỸ THUẬT LAMP Giới thiệu phương pháp LAMP Gần đây, một phương pháp nhân bản DNA mới đã được phát triển và giới thiệu ứng dụng, gọi là phương pháp LAMP (Loop-mediated isothermal amplification) tạo ra sản phẩm là DNA mạch vòm, nhìn thấy được bằng mắt thường để đánh giá kết quả (Notomi et al., 2000; Mori, Notomi, 2009). Trong vòng 10 năm qua kể từ khi được giới thiệu, LAMP đã thu hút sự quan tâm của các nhà nghiên cứu và xét nghiệm ở các cơ sở đối với tác nhân gây bệnh, do tính chính xác, đơn giản, hiệu suất, giá thành thấp và dễ thực hiện đánh giá. LAMP được phát triển thành bộ sinh phẩm (kit) thương mại phổ biến rộng rãi trên toàn thế giới (http://loopamp.eiken.co.jp/e/). Cho đến nay, đã có trên 350 công trình thực hiện LAMP, ứng dụng đối với hầu hết các loài (vi) sinh vật, ký sinh trùng gây bệnh ở người và động vật. Cùng tính đặc hiệu và tính nhạy như PCR, LAMP đã thực sự sử dụng như một phương pháp chẩn đoán tiên tiến với mọi đối tượng và trở nên cạnh tranh đắc lực với PCR, do tính nhanh chóng dễ làm, dễ đánh giá và không tốn kém nguyên vật liệu và bệnh phẩm (Nagamine et al., 2002; Parida, 2008; Mori, Notomi, 2009). Vậy phương pháp LAMP là gì? LAMP, tạm gọi là tổng hợp DNA-vòm (hay vắn tắt là kỹ thuật/phương pháp DNA-vòm) được phát triển năm 2000, là phương pháp tổng hợp DNA bằng 4 mồi đặc hiệu nhận biết 6 vùng khác biệt trên một gen đích từ một nguồn khuôn duy nhất, tạo nên một dạng sản phẩm DNA có cấu trúc vòm (stem-loop structure), hiển thị dưới dạng hỗn hợp vẩn đục màu trắng, nên dễ dàng phát hiện bằng mắt thường; nếu cho thêm chất huỳnh quang, sản phẩm sẽ phát quang dễ nhận biết hơn (Notomi et al., 2000; Parida et al., 2008) (Hình 2-3). Dạng vẩn đục màu trắng chính là Magnesium pyrophosphate (Mg2P2O7) một sản phẩm phụ của phản ứng sinh ra trong quá trình tạo DNA mạch vòm rất dễ quan sát đánh giá kết quả (Mori et al., 2001). Phương pháp DNA-vòm không đòi hỏi điều kiện phòng thí nghiệm hiện đại, nhân lực trình độ cao, phản ứng xảy ra đặc hiệu, thực hiện nhanh chóng (Mori, Notomi, 2009), rất thích hợp cho chẩn đoán phân tử virus, vi khuẩn và ký sinh trùng gây bệnh ở các nước đang phát triển vùng nhiệt đới (Dong et al., 2008; Parida, 2008). Nếu như PCR/RT-PCR, bao gồm cả multiplex-PCR, cho sản phẩm đặc hiệu là DNA mạch thẳng thuận lợi cho các phương pháp sinh học phân tử phân tích đặc tính của gen/hệ gen sau khi tách dòng và giải trình tự (Powledge, 2004; Ishmael, Stellato, 2008); thì LAMP/RT-LAMP cho sản phẩm đặc hiệu là DNA bắt cặp các chuỗi lại với nhau tạo mạch vòm không thuận lợi cho tách dòng và xem xét thành phần gen/hệ gen, nhưng DNA-vòm có lợi thế cực kỳ to lớn là sản phẩm hiển thị ở dạng vẩn đục trắng do sản sinh muối pyrophosphate (Mg2P2O7) hoặc với chất phát quang nhìn thấy bằng mắt thường, phục vụ nhanh chóng cho chẩn đoán (Mori et al., 2001;Goto et al., 2009) (Hình 3). Trong trường hợp LAMP sử dụng làm kit chẩn đoán, người ta thường thiết kế thêm một cặp mồi đặc hiệu bám vào hai đầu biên của vùng gen đích để thực hiện phản ứng PCR/RT-PCR, thu sản phẩm DNA-thẳng, rồi tách dòng, giải trình tự để kiểm nghiệm tính đặc hiệu cho chắc chắn (Parida, 2008). Phương pháp kiểm tra giả nghiệm cũng được thực hiện với các nguồn khuôn khác nhau của (vi) sinh vật cùng giống (genus) để đảm bảo chắc chắn tính chính xác và đặc hiệu của kit chẩn đoán LAMP (Parida, 2008; Parida et al., 2008). Nguyên lý kỹ thuật của LAMP Nguyên tắc thiết kế mồi cho LAMP Vùng gen đích cho các mồi của LAMP hoạt động không cần dài, chỉ khoảng vài trăm nucleotide, do vậy, không những thích hợp cho gen/hệ gen sinh vật bậc thấp như virus, vi khuẩn hoặc hệ gen ty thể có cấu trúc gen hoàn chỉnh (mono-cistronic gene), mà còn thích hợp với một exon của một cấu trúc gen sinh vật bậc cao (poly-cistronic gene), có độ dài khoảng vài trăm nucleotide là đủ (Goto et al., 2009). LAMP sử dụng 4 chuỗi mồi đặc hiệu được thiết kế hết sức cẩn thận và bảo đảm tính đặc hiệu cao, bám vào 6 chuỗi nucleotide của gen đích. Để bảo đảm tính thống nhất, tên chuỗi nucleotide của gen đích và tên mồi của LAMP được chính thức danh pháp hoá (Notomi et al., 2000; Mori, Notomi, 2001). Vùng gen đích: Sáu vùng gen đích được ký hiệu là F3, F2, F1 và B3, B2, B1 trên sợi DNA theo chiều 5’ -> 3’; do vậy, chuỗi nucleotide đối xứng bổ sung của 6 chuỗi này được ký hiệu là F3c, F2c, F1c và B3c, B2c, B1c trên sợi DNA theo chiều 3’ -> 5’. Vùng DNA của gen đích nằm giới hạn giữa F1 và B1 (Hình 2A). Mồi của LAMP: Mồi của LAMP gồm 4 chuỗi mồi đặc hiệu đó là FIP, BIP, F3, B3. Cụ thể: i) Mồi FIP (Forward Inner Primer) (chuỗi F1c + F2): Mồi xuôi bên trong có thành phần nucleotide chứa toàn bộ chuỗi F2 ở đầu 3’ đối xứng bổ sung (complementary) với toàn bộ chuỗi F2c, và được nối với chuỗi nucleotide của F1c tại đầu 5’, thực chất là chuỗi mồi kép, ghép chéo 2 chuỗi mồi đơn F1c + F2; ii) Mồi F3 (Forward Outer Primer): Mồi xuôi bên ngoài là chuỗi F3 đối xứng bổ sung với F3c; iii) Mồi BIP (Backward Inner Primer) (chuỗi B1c + B2): Mồi ngược bên trong có thành phần nucleotide chứa toàn bộ chuỗi B2 ở đầu 3’ đối xứng bổ sung (complementary) với toàn bộ chuỗi B2c, và được nối với chuỗi nucleotide của B1c tại đầu 5’, tương tự, thực chất là chuỗi mồi kép, ghép chéo 2 chuỗi mồi đơn B1c + B2; iv) Mồi B3 (Backward Outer Primer): Mồi ngược bên ngoài là chuỗi B3 đối xứng bổ sung với B3c (Hình 2A). Cơ chế hoạt động của LAMP LAMP là phản ứng tổng hợp DNA không phụ thuộc chu kỳ nhiệt độ (luôn luôn ở nhiệt độ 63oC), bao hàm kỹ thuật nhân bản DNA từ vùng gen đích của khuôn DNA/RNA trong một ống và một bước (one-tube/one-step), với sự xúc tác của enzyme Bst DNA polymerase, có độ nhạy và độ đặc hiệu cực kỳ cao, sản phẩm được tạo ra chỉ trong vòng 1 giờ, hiển thị dưới dạng muối Magnesium pyrophosphate ở dạng vẩn đục màu trắng (Hình3). LAMP là phản ứng nhân bản DNA hết sức đặc hiệu, chỉ xảy ra khi có điều kiện cần và đủ cho phản ứng, đó là chỉ khi cả 4 chuỗi mồi, bao gồm các mồi đơn F3, B3, các mồi chéo kép FIP (F1c+F2), BIP (B1c+B2) bám được vào 6 chuỗi đích của khuôn, thì sản phẩm DNA-vòm mới được tạo ra và mới phát hiện được. Chỉ môt hay vài mồi không hoạt động, hoặc không bám đặc hiệu, sản phẩm DNA-vòm sẽ không được tạo ra (Hình 2). Hình 2. Sơ đồ minh hoạ vùng gen đích, các mồi đặc hiệu của LAMP (A) và hoạt động của phản ứng qua các giai đoạn (B, C, D) tạo nên sản phẩm DNA cấu trúc vòm (E). Ghi chú: A: Mồi xuôi bên trong FIP chứa chuỗi nucleotide F1c và F2; mồi ngược bên trong BIP chứa chuỗi nucleotide B1c và B2; mồi xuôi bên ngoài F3 chứa chuỗi F3; mồi ngược B3 chứa chuỗi B3 được thiết kế để bám vào vùng gen đích tạo sản phẩm DNA có cấu trúc vòm. DNA cấu trúc vòm của sản phẩm được tạo ra từ DNA vùng gen đích giữa F1c (B1c) và F2c (B2c); B, C, D, E:Các giai đoạn mồi bám tiến hành tổng hợp DNA mạch thẳng trước và tiếp tục tạo vòm tổng hợp DNA mạch vòm làm sản phẩm. (Nguồn: http://loopamp.eiken.co.jp/e/lamp/principle.html). Hình 3. Hình ảnh hiển thị sản phẩm DNA-vòm của phản ứng LAMP với phát hiện bằng mắt thường ở dạng vẩn đục màu trắng (turbidity) (A) và bằng huỳnh quang khi sử dụng cho thêm hóa chất calcein (B). Ghi chú: A: Vẩn đục màu trắng là dương tính, trong suốt là âm tính; B: Phát sáng màu xanh và đục là dương tính, trong suốt hơi gợn xanh là âm tính. (Nguồn: Mori, Notomi, 2009). Cơ chế của phản ứng DNA-vòm có thể lược tóm trong 3 giai đoạn chính, bao gồm: giai đoạn khởi đầu (tạo khuôn, không chu kỳ), giai đoạn tổng hợp (tạo vòm, theo chu kỳ) và giai đoạn kết thúc (tạo sản phẩm) (Notomi et al., 2000) (Hình 2-BCDE). Các mồi đơn F3, B3 và một nửa mồi chéo kép FIP/BIP bắt đầu bám vào các vị trí tương ứng trên khuôn DNA (Hình 2A) và tiến hành tổng hợp sợi DNA bổ sung và kết thúc giai đoạn không chu kỳ tao khuôn; lúc này, phần nửa bên kia của mồi chéo kép lại bám ngược vào khuôn DNA và tổng hợp vòng trở lại sợi DNA bổ sung của nó (Hình 2B). Cứ như vậy, các mồi chéo kép của cả hai sợi khuôn luân hồi tạo vòm và tổng hợp DNA kết dính với nhau bằng vòm (stem-loop) ở cuối mỗi sợi (Hình 2CD). Phản ứng cứ vậy tiếp tục và có thể tạo nên 109 –1010 bản sao DNA mạch vòm (Hình 2E) trong vòng khoảng 15 phút đến 1 giờ. Điều đặc biệt là, khi sử dụng dẫn chất deoxy-nucleotide triphosphate (dNTP), theo qui luật tổng hợp DNA, các gốc phosphate được giải phóng ra sẽ kết hợp với i-on Mg++ có trong thành phần của phản ứng để tạo nên chất tủa vẩn đục Mg2P2O7 (Magnesium pyrophosphate) có màu trắng. Từ nguồn khuôn DNA/RNA khoảng vài picogram (pg), thậm chí vài femtogram (fg)) DNA là đủ (1 microgram = 103 nanogram = 106 picogram = 109 femtogram) đã có khoảng vài trăm ngàn chuỗi DNA vòm sinh ra đủ để tạo nên chất vẩn đục màu trắng của phản ứng dương tính. Có bao nhiêu chuỗi nucleotide ban đầu làm khuôn, kết quả sẽ có một hàm lượng chừng ấy sản phẩm tương ứng được tạo ra theo tỷ lệ thuận và nếu chuẩn hóa thành phần MgSO4 để biết lượng i-on Mg++, chúng ta có thể xác định được hàm lượng sản phẩm DNA vòm; và đó cũng là nguyên lý định lượng của phản ứng LAMP định lượng (real-time LAMP) (Goto et al., 2007). Cũng như ở PCR, khi khuôn có bản chất là RNA, đó là RNA thông tin (mRNA) hay RNA hệ gen (của virus RNA chẳng hạn) (gRNA) thì phản ứng cần thêm một bước chuyển đổi gọi là PCR ngược (RT-PCR); thì ở LAMP, cũng có thể thực hiện được LAMP ngược (RT-LAMP) và chỉ cần thêm vào phản ứng thành phần enzyme sao chép ngược (reverse transcriptase) hoặc chuyển cDNA (DNA bổ sung) trước, rồi thực hiện LAMP sau (Parida et al., 2008). Tương tự, cũng giống như phương pháp PCR định lượng (real-time PCR) dựa vào hàm lượng phát quang (fluorescence) sinh ra tương ứng với sản phẩm có từ khuôn; LAMP cũng được phát triển thành LAMP định lượng (real-time LAMP) trên cơ sở xác định hàm lượng chất tủa (turbidity), đo được bằng một dụng cụ gọi là vẩn kế (turbidimeter) có tỷ lệ thuận với sản phẩm DNA-vòm sản sinh ra trong phản ứng (Mori et al., 2004;Parida, 2008). Để khẳng định gen đích, người ta thường thiết kế thêm một cặp mồi đặc hiệu, ký hiệu LF và LB (loop forward và loop back) ở hai đầu biên của vùng gen đích để thực hiện phản ứng PCR/(RT-PCR), nâng tổng số chuỗi mồi tất cả là 6, nhằm thu được đoạn DNA-thẳng của vùng gen đích để biết chính xác sản phẩm cần chẩn đoán. Như vậy, hỗn hợp mồi LAMP hoặc bao gồm 4 chuỗi mồi nhận biết 6 chuỗi gen đích, chỉ để thực hiện LAMP/RT-LAMP thông thường hoặc 6 chuỗi mồi đặc hiệu nhận biết 8 chuỗi gen đích, để thực hiện LAMP/RT-LAMP và kết hợp với PCR (Parida, 2008; Mori, Notomi, 2009). LAMP ứng dụng trong chẩn đoán phân tử xác định và phân biệt ký sinh trùng Kể từ khi được giới thiệu từ những năm 2000, càng ngày càng có nhiều nghiên cứu đưa LAMP vào ứng dụng chẩn đoán ở các lĩnh vực khác nhau, trong đó có ký sinh trùng. Đối với ký sinh trùng, nhiều khi chúng cùng tồn tại ở một vị trí nên rất dễ lấy bệnh phẩm, tuy nhiên việc phân biệt trong chẩn đoán và xác nhận sự có mặt của loài gây bệnh là không dễ dàng khi sử dụng phương pháp hình thái học. Việc phát hiện đặc hiệu loài gây bệnh để phân biệt được nhiều loài khác càng nhanh càng tốt có ý nghĩa rất lớn trong chẩn đoán, do vậy, LAMP trở nên một công cụ hữu hiệu xác định nhanh đối tượng trong số đông cùng tồn tại (Criado-Fornelio, 2007; Adams, Hamilton, 2008). Trong 10 năm qua, bên cạnh việc thăm dò phương pháp và xây dựng bộ kit với tiêu chuẩn chẩn đoán sau khi khảo nghiệm, LAMP đã được ứng dụng trong thực tế với nhiều loại ký sinh trùng khcá nhau gây bệnh ở động vật và người. LAMP đã được xây dựng để ứng dụng chẩn đoán phân biệt các loại ký sinh trùng đơn bào, trước hết đó là các loài Trypanosoma spp gây bệnh ở châu Phi (Kuboki et al., 2003; Adams, Hamilton, 2008; Thekisoe et al., 2009), bao gồm chẩn đoán phân biệt các loài trong phân giống Trypanozoon (Njiru et al., 2009) hoặc chính xác xác định loài Trypanosoma brucei rhodesiense (Njiru et al., 2008). LAMP cũng được thăm dò chẩn đoán ký sinh trùng đường máu ở động vật là Trypanosoma evansi qua kiểm tra trên lợn thực nghiệm (Thekisoe et al., 2005; 2007). Một số loại ký sinh trùng đường máu khác cũng được ứng dụng LAMP để chẩn đoán trong đó có lê dạng trùng Babesia orientalisgây bệnh ở trâu ở Trung Quốc (He et al., 2009) hay phát hiện loài Babesia gibsoni ở Nhật Bản (Ikadai et al., 2004); hoặc phân biệt các loài trong giống Babesia (Guan et al., 2008) hay các loài piroplasma bao gồm Babesia spp và Theileria spp gây bệnh ở bò (Criado-Fornelio, 2007; Thekisoe et al., 2007; Liu et al., 2008; Salih et al., 2008), đặc biệt là loài Theileria parva gây bệnh ở bò (Thekisoe et al., 2010). Đối với Leishmania donovani, Takagi et al. (2009), phương pháp LAMP chẩn đoán rất nhạy chỉ cần 1 fg khuôn DNA là đủ phát hiện và phân biệt với các loài Leishmania spp (L. infantum, L. major, L. mexicana, L. tropica, L. braziliensis) khác. Phương pháp LAMP cũng phát hiện ký sinh trùng đơn bào đường máu Toxoplasma gondii từ đất; và loài đơn bào gây bệnh sang người này đã được một số nhà nghiên cứu lấy làm đối tượng xây dựng thành kit chẩn đoán trên người (Sotiriadou, Karanis, 2008; Krasteva et al., 2009; Zhang et al., 2009) Đơn bào gây bệnh người và động vật truyền qua thức ăn nước uống thuộc giống Cryptosporidium và Giardia được đặc biệt quan tâm sử dụng LAMP do tính nhạy của phương pháp này (Bakheit et al., 2009), trước hết là chẩn đoán Cryptosporidium spp và Giardia duodenalis trong nước uống (Inomata et al., 2009; Plutzer, Tomor, 2009; Karanis, 2010) và trong động vật truyền lây (Plutzer, Tomor, 2009), trong phân thải và môi trường (Karanis et al., 2007). Bên cạnh đó lỵ amíp Acanthamoeba (Lek-Uthai et al., 2009) hay Entamoeba histolytica (Liang et al., 2009) cũng là đối tượng của phương pháp chân đoán mới này. Một trong những ứng dụng có hiệu quả trong sử dụng LAMP đó là phát triện phương pháp chẩn đoán phân biệt 4 loài ký sinh trùng sốt rét Plasmodium spp (Han et al., 2007; Aonuma et al., 2008) hoặc đơn lẻ từng loài Plasmodium falciparum (Poon et al., 2005; Yamamura et al., 2009) và Plasmodium vivax (Chen et al., 2010). Đối với giun tròn, chỉ mới có một công trình nghiên cứu xây dựng phương pháp LAMP chẩn đoán giun chỉ Dirofilaria immitis từ vector là muỗi (Aonuma et al., 2009); và đối với sán dẹt, mới có loài sán máng Schistosoma japonicum (Xu et al., 2010) và các loài sán dây Taenia spp (Nkouawa et al., 2009) làm đối tượng của LAMP. Giun tròn và sán dẹt gây bệnh truyền từ động vật sang người còn có rất nhiều loài nguy hiểm khác cần được xây dựng phương pháp chẩn đoán nhanh bằng LAMP. VÀI LỜI KẾT LUẬN VÀ ĐỊNH HƯỚNG Ở VIỆT NAM Có rất nhiều phương pháp chẩn đoán (diagnosis), phát hiện (detection) và giám định (identification) tác nhân gây bệnh, trong đó có ký sinh trùng, mà cho đến gần đây, kỹ thuật nuôi cấy, xác định hình thái học bằng kính hiển vi quang học và gây bệnh thực nghiệm vẫn là những phương pháp cổ điển được ứng dụng. Nhiều kỹ thuật nền và dựa vào đó là những phương pháp mới, nhanh, nhạy, chính xác, kỹ thuật cao, phần lớn là nhân bản và phát hiện DNA của ký sinh trùng gây bệnh, đã được phát triển và ứng dụng trong vòng 20 năm qua. Từ những kỹ thuật nền như thế này, các bộ kit chẩn đoán đã được thương mại hóa phát huy tác dụng hỗ trợ ở các cơ sở nghiên cứu và bệnh viện, khẳng định lợi thế hơn hẳn của chẩn đoán phân tử khi kết hợp hoặc sử dụng riêng so với các phương pháp chẩn đoán thông thường khác. Các phương pháp nhân bản DNA sử dụng đa mồi trước hết là Multiplex-PCR và LAMP đã và đang được ứng dụng rộng rãi do tính tiện lợi và khả năng phát hiện, chẩn đoán chính xác đối tượng, đặc biệt là đối với ký sinh trùng một loại tác nhân gây bệnh có chu trình sống đặc biệt qua nhiều giai đoạn phát triển, nhiều loài vật chủ và khu trú nhiều vị trí khác nhau. Trên thế giới, đã có hàng trăm công trình multiplex-PCR và hàng chục công trình LAMP được xây dựng ứng dụng trong lĩnh vực chẩn đoán phân tử bằng phương pháp tổng hợp DNA sản phẩm đa mồi để xác định và phân biệt ký sinh trùng. Tại Việt Nam, tuy chưa nhiều, nhưng đã có những định hướng ứng dụng phương pháp tổng hợp DNA sản phẩm đa mồi trong nghiên cứu chẩn đoán ký sinh trùng và đã thu được một số kết quả nhất định (xem Nguyễn Văn Đề, Lê Thanh Hòa, 2010). Chúng tôi không có ý định giới thiệu tổng quan kết quả nghiên cứu tại Việt Nam ở trong bài báo này. Việt Nam là một nước nhiệt đới có môi trường thuận lợi cho các loại ký sinh trùng phát triển, truyền lây và gây bệnh giữa môi trường, động vật và người. Hơn nữa, mặt bằng vệ sinh đời sống chưa được đảm bảo, con người và động vật dễ nhiễm hoặc/và tái nhiễm ký sinh trùng nguy hiểm, nên việc nghiên cứu ứng dụng các phương pháp chẩn đoán nhanh và chính xác có tầm quan trọng đặc biệt. Chưa có công bố kết quả nghiên cứu ứng dụng LAMP đối với ký sing trùng tại Việt Nam. Tuy nhiên, chúng tôi đã xây dựng xong phương pháp LAMP và đang thực hiện với một số loài sán lá gây bệnh thường gặp, sau đó tiến hành hoàn thiện Kit chẩn đoán LAMP để chuyển giao cho các cơ sở có nhu cầu, vì đây là một phương pháp đơn giản và tiện lợi. Các phương pháp và bộ kit chẩn đoán phân tử cũng đang được xây dựng và hoàn thiện. Chúng tôi hy vọng rằng, với những kỹ thuật chẩn đoán phân tử hiện đại như Multiplex-PCR và LAMP, các cơ sở nghiên cứu và triển khai xét nghiệm trong cả nước sẽ có điều kiện xác định nhanh, dễ dàng và chính xác tác nhân gây bệnh ký sinh trùng góp phần giảm thiểu bệnh nâng cao sức khoẻ cộng đồng. Lời cảm ơn:Chúng tôi cảm ơn Bộ Y tế hỗ trợ kinh phí cho đề tài cấp Bộ: “Sử dụng phương pháp phân tử xây dựng kit để chẩn đoán các loài ký sinh trùng chủ yếu ở Việt Nam” (2010 -2011) do PGS.TS. Lê Thanh Hòa chủ nhiệm, để hoàn thành công trình này. TÀI LIỆU THAM KHẢO
1.Adams ER, Hamilton PB (2008) New molecular tools for the identification of trypanosome species. Future Microbiol 3: 167-176. Review.
2.Ajzenberg D, Dumètre A, Dardé ML (2005) Multiplex PCR for typing strains of Toxoplasma gondii. J Clin Microbiol 43(4): 1940-1943.
3.Alhassan A, Pumidonming W, Okamura M, Hirata H, Battsetseg B, Fujisaki K, Yokoyama N, Igarashi I (2005) Development of a single-round and multiplex PCR method for the simultaneous detection of Babesia caballi and Babesia equi in horse blood. Vet Parasitol 129(1-2): 43-49.
4.Aonuma H, Suzuki M, Iseki H, Perera N, Nelson B, Igarashi I, Yagi T, Kanuka H, Fukumoto S (2008) Rapid identification of Plasmodium-carrying mosquitoes using loop-mediated isothermal amplification. Biochem Biophys Res Commun 376(4): 671-676.
5.Aonuma H, Yoshimura A, Perera N, Shinzawa N, Bando H, Oshiro S, Nelson B, Fukumoto S, Kanuka H (2009) Loop-mediated isothermal amplification applied to filarial parasites detection in the mosquito vectors: Dirofilaria immitis as a study model. Parasit Vectors 2(1):15.
6.Bakheit MA, Torra D, Palomino LA, Thekisoe OM, Mbati PA, ongerth J, KaranisP (2008) Sensitive and specific detection of Cryptosporidium species in PCR-negative samples by loop-mediated isothermal DNA amplification and confirmation of generated LAMP products by sequencing. Vet Parasitol 158(1-2): 11-22.
7.Chandler HL, Colitz CM (2006) Molecular biology for the clinician: understanding current methods. J Am Anim Hosp Assoc. 42(5): 326-335. Review.
8.Chen JH, Lu F, Lim CS, Kim JY, Ahn HJ, Suh IB, Takeo S, Tsuboi T, Sattabongkot J, Han ET (2010) Detection of Plasmodium vivax infection in the Republic of Korea by loop-mediated isothermal amplification (LAMP). Acta Trop 113(1): 61-65.
9.Coiras MT, Aguilar JC, García ML, Casas I, Pérez-Breña P (2004) Simultaneous detection of fourteen respiratory viruses in clinical specimens by two multiplex reverse transcription nested-PCR assays. J Med Virol 72(3): 484-495.
10.Criado-Fornelio A (2007) A review of nucleic-acid-based diagnostic tests for Babesia and Theileria, with emphasis on bovine piroplasms. Parasitologia 49 Suppl 1: 39-44.
11.Davidson RK, Oines O, Madslien K, Mathis A (2009) Echinococcus multilocularis-adaptation of a worm egg isolation procedure coupled with a multiplex PCR assay to carry out large-scale screening of red foxes (Vulpes vulpes) in Norway. Parasitol Res 104(3): 509-514.
12.De Lellis L, Curia MC, Veschi S, Aceto GM, Morgano A, Cama A (2008) Methods for routine diagnosis of genomic rearrangements: multiplex PCR-based methods and future perspectives. Expert Rev Mol Diagn 8(1): 41-52. Review.
13.de Pita-Pereira D, Cardoso MA, Alves CR, Brazil RP, Britto C (2008) Detection of natural infection in Lutzomyia cruzi and Lutzomyia forattinii (Diptera: Psychodidae: Phlebotominae) by Leishmania infantum chagasi in an endemic area of visceral leishmaniasis in Brazil using a PCR multiplex assay. Acta Trop 107(1): 66-69.
14.Deng HW, Zhou Y, Recker RR, Johnson ML, Li J (2000) Fragment size difference between multiplex and singleplex PCR products and their practical implications. Biotechniques 29(2): 298-304; 307-308.
15.Doanh PN, Shinohara A, Horii Y, Habe S, Nawa Y (2009). Discovery of Paragonimus westermani in Vietnam and its molecular phylogenetic status in P. westermani complex. Parasitol Res. 104(5):1149-55.
16.Doanh PN, Shinohara A, Horii Y, Habe S, Nawa Y, Le NT (2007). Description of a new lung fluke species, Paragonimus vietnamensis sp. nov. (Trematoda, Paragonimidae), found in northern Vietnam. Parasitol Res 101(6):1495-501.
17.Doanh PN, Shinohara A, Yoichiro Horii Y, Shigehisa Habe S, Yukifumi Nawa Y and Nguyen Thi Le NT (2008). Discovery of Paragonimus proliferus in Northern Vietnam and their molecular phylogenetic status among genus Paragonimus. Parasitol Res 102:677–683.
18.Dong J, Olano JP, McBride JW, Walker DH (2008) Emerging pathogens: challenges and successes of molecular diagnostics. J Mol Diagn 10: 185–197.
19.Edwards MC, Gibbs RA (1994) Multiplex PCR: advantages, development, and applications. PCR Methods Appl 3(4): S65-75. Review.
20.Elnifro EM, Ashshi AM, Cooper RJ, Klapper PE (2000) Multiplex PCR: optimization and application in diagnostic virology. Clin Microbiol Rev 13(4): 559-570. Review.
21.Evangelopoulos A, Spanakos G, Patsoula E, Vakalis N, Legakis N (2000) A nested, multiplex, PCR assay for the simultaneous detection and differentiation of Entamoeba histolytica and Entamoeba dispar in faeces. Ann Trop Med Parasitol 94(3): 233-240.
22.Fernandez S, Pagotto AH, Furtado MM, Katsuyama AM, Madeira AM, Gruber A (2003) A multiplex PCR assay for the simultaneous detection and discrimination of the seven Eimeria species that infect domestic fowl. Parasitology 127(4): 317-325.
23.Fernandez S, Pagotto AH, Furtado MM, Katsuyama AM, Madeira AM, Gruber A (2003) A multiplex PCR assay for the simultaneous detection and discrimination of the seven Eimeria species that infect domestic fowl. Parasitology 127(4): 317-325.
24.Galluzzi L, Magnani M, Saunders N, Harms C, Bruce IJ (2007) Current molecular techniques for the detection of microbial pathogens. Sci Prog 90(1): 29-50. Review.
25.Gilbert GL (2002) Molecular diagnostics in infectious diseases and public health microbiology: cottage industry to postgenomics. Trends Mol Med 8: 280-287.
26.Gile M, Warhurst DC, Webster KA, West DM, Marshall JA (2002) A multiplex allele specific polymerase chain reaction (MAS-PCR) on the dihydrofolate reductase gene for the detection of Cryptosporidium parvum genotypes 1 and 2. Parasitology 125(1): 35-44.
27.Gill P, Ghaemi A (2008) Nucleic acid isothermal amplification technologies: a review. Nucleosides Nucleotides Nucleic Acids 27(3): 224-243. Review.
28.González LM, Montero E, Harrison LJ, Parkhouse RM, Garate T (2000) Differential diagnosis of Taenia saginata and Taenia solium infection by PCR. J Clin Microbiol 38(2): 737-744.
29.González LM, Montero E, Morakote N, Puente S, Díaz De Tuesta JL, Serra T, López-Velez R, McManus DP, Harrison LJ, Parkhouse RM, Gárate T (2004) Differential diagnosis of Taenia saginata and Taenia saginata asiatica taeniasis through PCR. Diagn Microbiol Infect Dis 49(3):183-188.
30.Goto M, Honda E, Ogura A, Nomoto A, Hanaki K (2009) Colorimetric detection of loop-mediated isothermal amplification reaction by using hydroxy naphthol blue. Biotechniques 46(3): 167-172.
31.Guan G, Chauvin A, Luo J, Inoue N, Moreau E, Liu Z, Gao J, Thekisoe OM, Ma M, Liu A, Dang Z, Liu J, Ren Q, Jin Y, Sugimoto C, Yin H (2008) The development and evaluation of a loop-mediated isothermal amplification (LAMP) method for detection of Babesia spp. infective to sheep and goats in China. Exp Parasitol 120(1): 39-44.
32.Guy RA, Payment P, Krull UJ, Horgen PA (2003) Real-time PCR for quantification of Giardia and Cryptosporidium in environmental water samples and sewage. Appl Environ Microbiol 69(9): 5178-85.
33.Han ET, Watanabe R, Sattabongkot J, Khuntirat B, Sirichaisinthop J, Iriko H, Jin L, Takeo S, Tsuboi T (2007) Detection of four Plasmodium species by genus- and species-specific loop-mediated isothermal amplification for clinical diagnosis. J Clin Microbiol 45(8): 2521-2528.
34.Haque R, Roy S, Siddique A, Mondal U, Rahman SM, Mondal D, Houpt E, Petri WA Jr (2007) Multiplex real-time PCR assay for detection of Entamoeba histolytica, Giardia intestinalis, and Cryptosporidium spp. Am J Trop Med Hyg 76(4): 713-717.
35.Harris E, Kropp G, Belli A, Rodriguez B, Agabian N (1998) Single-step multiplex PCR assay for characterization of New World Leishmania complexes. J Clin Microbiol 36(7): 1989-1995.
36.He L, Zhou YQ, Oosthuizen MC, Zhao JL (2009) Loop-mediated isothermal amplification (LAMP) detection of Babesia orientalis in water buffalo (Bubalus babalis, Linnaeus, 1758) in China. Vet Parasitol 165(1-2): 36-40.
37.Hu M, Gasser RB (2006) Mitochondrial genomes of parasitic nematodes-progress and perspectives. Trends Parasitol 22(2): 78-84. Review.
38.Ikadai H, Tanaka H, Shibahara N, Matsuu A, Uechi M, Itoh N, Oshiro S, Kudo N, Igarashi I, Oyamada T (2004) Molecular evidence of infections with Babesia gibsoni parasites in Japan and evaluation of the diagnostic potential of a loop-mediatedisothermal amplification method. J Clin Microbiol 42(6): 2465-2469.
39.Inomata A, Kishida N, Momoda T, Akiba M, Izumiyama S, Yagita K, Endo T (2009) Development and evaluation of a reverse transcription-loop-mediated isothermal amplification assay for rapid and high-sensitive detection of Cryptosporidium in water samples. Water Sci Technol 60(8): 2167-2172.
40.Ishmael FT, Stellato C (2008) Principles and applications of polymerase chain reaction: basic science for the practicing physician. Ann Allergy Asthma Immunol 101(4): 437-443.
41.Jorquera A, González R, Marchán-Marcano E, Oviedo M, Matos M (2005) Multiplex-PCR for detection of natural Leishmania infection in Lutzomyia spp. captured in an endemic region for cutaneous leishmaniasis in state of Sucre, Venezuela. Mem Inst Oswaldo Cruz 100(1): 45-48.
42.Kaderali L (2007) Primer design for multiplexed genotyping. Methods Mol Biol 402: 269-286. Review.
43.Karanis P, Thekisoe O, Kiouptsi K, ongerth J, Igarashi I, Inoue N (2007) Development and preliminary evaluation of a loop-mediated isothermal amplification procedure for sensitive detection of Cryptosporidium oocysts in fecal and water samples. Appl Environ Microbiol 73(17): 5660-5662.
44.Khairnar K, Parija SC (2007) A novel nested multiplex polymerase chain reaction (PCR) assay for differential detection of Entamoeba histolytica, E. moshkovskii and E. dispar DNA in stool samples. BMC Microbiol 7: 47.
45.Krasteva D, Toubiana M, Hartati S, Kusumawati A, Dubremetz JF, Widada JS (2009) Development of loop-mediated isothermal amplification (LAMP) as a diagnostic tool of toxoplasmosis. Vet Parasitol 162(3-4): 327-331.
46.Kubista M, Andrade JM, Bengtsson M, Forootan A, Jonák J, Lind K, Sindelka R, Sjöback R, Sjögreen B, Strömbom L, Ståhlberg A, Zoric N (2006) The real-time polymerase chain reaction. Mol Aspects Med 27(2-3): 95-125. Review.
47.Kuboki N, Inoue N, Sakurai T, Di Cello F, Grab DJ, Suzuki H, Sugimoto C, Igarashi I (2003) Loop-mediated isothermal amplification for detection of African trypanosomes. J Clin Microbiol 41(12): 5517-5524.
48.Lardeux F, Tejerina R, Aliaga C, Ursic-Bedoya R, Lowenberger C, Chavez T (2008) Optimization of a semi-nested multiplex PCR to identify Plasmodium parasites in wild-caught Anopheles in Bolivia, and its application to field epidemiological studies. Trans R Soc Trop Med Hyg 102(5): 485-492.
49.Le TH, Blair D and McManus DP (2002) Mitochondrial genomes of parasitic flatworms. Trends Parasitol 18: 206-213.
50.Le TH, De NV, Blair D, Sithithaworn P and McManus DP (2006) Clonorchis sinensis and Opisthorchis viverrini: development of a mitochondrial-based multiplex PCR for their identification and discrimination. Experimental Parasitology, 112(2): 109-114.
51.Lê Thanh Hòa (2007). Chẩn đoán phân loại ký sinh trùng bằng các phương pháp truyền thống và sinh học phân tử. Tạp chí Y học thành phố Hồ Chí Minh, 11(PB2):1-8.
52.Lê Thanh Hòa (2009). Sử dụng phương pháp phân tử xây dựng kit để chẩn đoán các loài ký sinh trùng chủ yếu ở Việt Nam. Đề tài cấp Bộ Y tế (2010-2011).
53.Lê Thanh Hoà, Nguyễn Văn Đề, Nguyễn Bích Nga, Nguyễn Quốc Doanh, Lê Đình Công (2002). Phát hiện loài sán dây Taenia asiatica ký sinh trên người ở Việt Nam bằng phương pháp giám định sinh học phân tử. Tạp chí Sinh học, 24(1):23-29.
54.Lê Thanh Hòa, Trương Nam Hải (2010). Phương pháp tổng hợp DNA sản phẩm đa mồi (multiplex-PCR và LAMP) trong chẩn đoán phân tử để xác định và phân biệt tác nhân gây bệnh. Tổng quan. Tạp chí Công nghệ Sinh học (đang in).
55.Lek-Uthai U, Passara R, Roongruangchai K, Buddhirakkul P, Thammapalerd N (2009) Rapid identification of Acanthamoeba from contact lens case using loop-mediated isothermal amplification method. Exp Parasitol 121(4): 342-345.
56.Liang SY, Chan YH, Hsia KT, Lee JL, Kuo MC, Hwa KY, Chan CW, Chiang TY, Chen JS, Wu FT, Ji DD (2009) Development of loop-mediated isothermal amplification assay for detection of Entamoeba histolytica. J Clin Microbiol 47(6): 1892-1895.
57.Lin RQ, Ai L, Zou FC, Verweij JJ, Jiang Q, Li MW, Song HQ, Zhu XQ (2008) A multiplex PCR tool for the specific identification of Oesophagostomum spp. from pigs. Parasitol Res 103(4): 993-997.
58.Littlewood DT (2008) Platyhelminth systematics and the emergence of new characters. Parasite 15(3): 333-341.
59.Liu Z, Hou J, Bakheit MA, Salih DA, Luo J, Yin H, Ahmed JS, Seitzer U (2008) Development of loop-mediated isothermal amplification (LAMP) assay for rapid diagnosis of ovine theileriosis in China. Parasitol Res 103(6): 1407-1412.
60.Magalhães KG, Jannotti-Passos LK, Caldeira RL, Berne ME, Muller G, Carvalho OS, Lenzi HL (2008) Isolation and detection of Fasciola hepatica DNA in Lymnaea viatrix from formalin-fixed and paraffin-embedded tissues through multiplex-PCR. Vet Parasitol 152(3-4): 333-338.
61.Magalhães KG, Passos LK, Carvalho Odos S (2004) Detection of Lymnaea columella infection by Fasciola hepatica through Multiplex-PCR. Mem Inst Oswaldo Cruz 99(4): 421-424.
62.McCarthya J, Moore TA (2000) Emerging helminth zoonoses. Intl J Parasitol 30: 1351-1360.
63.Mishra K, Raj DK, Hazra RK, Dash AP, Supakar PC (2007) The development and evaluation of a single step multiplex PCR method for simultaneous detection of Brugia malayi and Wuchereria bancrofti. Mol Cell Probes 21(5-6): 355-362.
64.Mori Y, Kitao M, Tomita N, Notomi T (2004) Real-time turbidimetry of LAMP reaction for quantifying template DNA. J Biochem Biophys Methods 59(2): 145-157.
65.Mori Y, Nagamine K, Tomita N, Notomi T (2001) Detection of loop-mediated isothermal amplification reaction by turbidity derived from magnesium pyrophosphate formation. Biochem Biophys Res Commun 289(1): 150-154.
66.Mori Y, Notomi T (2009) Loop-mediated isothermal amplification (LAMP): a rapid, accurate, and cost-effective diagnostic method for infectious diseases. J Infect Chemother 15(2): 62-69. Review.
67.Nagamine K, Kuzuhara Y, Notomi T (2002) Isolation of single-stranded DNA from loop-mediated isothermal amplification products. Biochem Biophys Res Commun 290(4): 1195-1198.
68.Nguyễn Văn Đề, Lê Thanh Hoà (2006). Xác định loài Taenia saginata và Taenia asiatica gây bệnh trên người ở Việt Nam bằng sinh học phân tử. Tạp chí Y học Việt Nam, 318:42-50.
69.Nguyễn Văn Đề, Lê Thanh Hòa (2010). Sách: “Sán dây/Ấu trùng sán lợn và sinh học phân tử ứng dụng (Taenia/cysticercosis and molecular application)”. Nhà xuất bản Y học, Hà Nội (326 trang).
70.Njiru ZK, Mikosza AS, Armstrong T, Enyaru JC, Ndung'u JM, Thompson AR (2008) Loop-mediated isothermal amplification (LAMP) method for rapid detection of Trypanosoma brucei rhodesiense. PLoS Negl Trop Dis 2(1): e147.
71.Njiru ZK, Mikosza AS, Matovu E, Enyaru JC, Ouma JO, Kibona SN, Thompson RC, Ndung'u JM (2008) African trypanosomiasis: sensitive and rapid detection of the sub-genus Trypanozoon by loop-mediated isothermal amplification (LAMP) of parasite DNA. Int J Parasitol 38(5): 589-599.
72.Nkouawa A, Sako Y, Nakao M, Nakaya K, Ito A (2009) Loop-mediated isothermal amplification method for differentiation and rapid detection of Taenia species. J Clin Microbiol 47(1): 168-174.
73.Notomi T, Okayama H, Masubuchi H, Yonekawa T, Watanabe K, Amino N, Hase T (2000) Loop-mediated isothermal amplification of DNA. Nucleic Acids Res 28: E63.
74.Nowakowska D, Colón I, Remington JS, Grigg M, Golab E, Wilczynski J, Sibley LD (2006) Genotyping of Toxoplasma gondii by multiplex PCR and peptide-based serological testing of samples from infants in Poland diagnosed with congenital toxoplasmosis. J Clin Microbiol 44(4): 1382-1389.
75.Ono K, Satoh M, Yoshida T, Ozawa Y, Kohara A, Takeuchi M, Mizusawa H, Sawada H (2007) Species identification of animal cells by nested PCR targeted to mitochondrial DNA. In Vitro Cell Dev Biol Anim 43(5-6): 168-175.
76.Orlandi PA, Carter L, Brinker AM, da Silva AJ, Chu DM, Lampel KA, Monday SR (2003) Targeting single-nucleotide polymorphisms in the 18S rRNA gene to differentiate Cyclospora species from Eimeria species by multiplex PCR. Appl Environ Microbiol 69(8): 4806-4813.
77.Padley D, Moody AH, Chiodini PL, Saldanha J (2003) Use of a rapid, single-round, multiplex PCR to detect malarial parasites and identify the species present. Ann Trop Med Parasitol 97(2): 131-137.
78.Parida MM (2008) Rapid and real-time detection technologies for emerging viruses of biomedical importance. J Biosci 33(4): 617-628.
79.Parida MM, Sannarangaiah S, Dash PK, Rao PV, Morita K (2008) Loop mediated isothermal amplification (LAMP): a new generation of innovative gene amplification technique; perspectives in clinical diagnosis of infectious diseases. Rev Med Virol 18(6): 407-421. Review.
80.Patel S, Pedraza-Díaz S, McLauchlin J (1999) The identification of Cryptosporidium species and Cryptosporidium parvum directly from whole faeces by analysis of a multiplex PCR of the 18S rRNA gene and by PCR/RFLP of the Cryptosporidium outer wall protein (COWP) gene. Int J Parasitol 29(8): 1241-1247.
81.Patsoula E, Spanakos G, Sofianatou D, Parara M, Vakalis NC (2003) A single-step, PCR-based method for the detection and differentiation of Plasmodium vivax and P. falciparum. Ann Trop Med Parasitol 97(1): 15-21.
82.Plutzer J, Karanis P (2009) Rapid identification of Giardia duodenalis by loop-mediated isothermal amplification (LAMP) from faecal and environmental samples and comparative findings by PCR and real-time PCR methods. Parasitol Res 104(6): 1527-1533.
83.Plutzer J, Tomor B (2009) The role of aquatic birds in the environmental dissemination of human pathogenic Giardia duodenalis cysts and Cryptosporidium oocysts in Hungary. Parasitol Int 58(3): 227-231.
84.Plutzer J, Törökné A, Karanis P (2010) Combination of ARAD microfibre filtration and LAMP methodology for simple, rapid and cost-effective detection of human pathogenic Giardia duodenalis and Cryptosporidium spp. in drinking water. Lett Appl Microbiol 50(1): 82-88.
85.Poon LL, Wong BW, Ma EH, Chan KH, Chow LM, Abeyewickreme W, Tangpukdee N, Yuen KY, Guan Y, Looareesuwan S, Peiris JS (2005) Sensitive and inexpensive molecular test for falciparum malaria: detecting Plasmodium falciparum DNA directly from heat-treated blood by loop-mediated isothermal amplification. Clin Chem 52(2): 303-306.
86.Powledge TM (2004) The polymerase chain reaction. Adv Physiol Educ 28: 44–50.
87.Rao RU, Huang Y, Bockarie MJ, Susapu M, Laney SJ, Weil GJ (2009) A qPCR-based multiplex assay for the detection of Wuchereria bancrofti, Plasmodium falciparum and Plasmodium vivax DNA. Trans R Soc Trop Med Hyg 103(4): 365-370.
88.Rodríguez-González I, Marín C, Longoni SS, Mateo H, Alunda JM, Minaya G, Gutiérrez-Sánchez R, Vargas F, Sánchez-Moreno M (2007) Identification of New World Leishmania species from Peru by biochemical techniques and multiplex PCR assay. FEMS Microbiol Lett 267(1): 9-16.
89.Rougemont M, Van Saanen M, Sahli R, Hinrikson HP, Bille J, Jaton K (2004) Detection of four Plasmodium species in blood from humans by 18S rRNA gene subunit-based and species-specific real-time PCR assays. J Clin Microbiol 42(12): 5636-5643.
90.Salih DA, Liu Z, Bakheit MA, Ali AM, El Hussein AM, Unger H, Viljoen G, Seitzer U, Ahmed JS (2008) Development and evaluation of a loop-mediated isothermal amplification method for diagnosis of tropical theileriosis. Transbound Emerg Dis 55(5-6): 238-243.
91.Singh DV (2003) Hexaplex PCR for rapid detection of virulence factors. Expert Rev Mol Diagn 3(6): 781-784. Review.
92.Sotiriadou I, Karanis P (2008) Evaluation of loop-mediated isothermal amplification for detection of Toxoplasma gondii in water samples and comparative findings by polymerase chain reaction and immunofluorescence test (IFT). Diagn Microbiol Infect Dis 62(4): 357-365.
93.Sugiyama H, Morishima Y, Rangsiruji A, Binchai S, Ketudat P, Kawanaka M (2006) Application of multiplex PCR for species discrimination using individual metacercariae of Paragonimus occurring in Thailand. Southeast Asian J Trop Med Public Health 37 Suppl 3: 48-52.
94.Takagi H, Itoh M, Islam MZ, Razzaque A, Ekram AR, Hashighuchi Y, Noiri E, Kimura E (2009) Sensitive, specific, and rapid detection of Leishmania donovani DNA by loop-mediated isothermal amplification. Am J Trop Med Hyg 81(4): 578-582.
95.Thekisoe OM, Bazie RS, Coronel-Servian AM, Sugimoto C, Kawazu S, Inoue N (2009) Stability of Loop-Mediated Isothermal Amplification (LAMP) reagents and its amplification efficiency on crude trypanosome DNA templates. J Vet Med Sci 71(4): 471-475.
96.Thekisoe OM, Inoue N, Kuboki N, Tuntasuvan D, Bunnoy W, Borisutsuwan S, Igarashi I, Sugimoto C (2005) Evaluation of loop-mediated isothermal amplification (LAMP), PCR and parasitological tests for detection of Trypanosoma evansi in experimentally infected pigs. Vet Parasitol 130(3-4): 327-330.
97.Thekisoe OM, Omolo JD, Swai ES, Hayashida K, Zhang J, Sugimoto C, Inoue N (2007) Preliminary application and evaluation of loop-mediated isothermal amplification (LAMP) for detection of bovine theileriosis and trypanosomosis in Tanzania. onderstepoort J Vet Res 74(4): 339-342.
98.Thekisoe OM, Rambritch NE, Nakao R, Bazie RS, Mbati P, Namangala B, Malele I, Skilton RA, Jongejan F, Sugimoto C, Kawazu S, Inoue N (2010) Loop-mediated isothermal amplification (LAMP) assays for detection of Theileria parva infections targeting the PIM and p150 genes. Int J Parasitol 40(1): 55-61.
99.Trachsel D, Deplazes P, Mathis A (2007) Identification of taeniid eggs in the faeces from carnivores based on multiplex PCR using targets in mitochondrial DNA. Parasitology 134(6): 911-920
100.Veiga MI, Ferreira PE, Björkman A, Gil JP (2006) Multiplex PCR-RFLP methods for pfcrt, pfmdr1 and pfdhfr mutations in Plasmodium falciparum. Mol Cell Probes 20(2): 100-104.
101.Verweij JJ, Brienen EA, Ziem J, Yelifari L, Polderman AM, Van Lieshout L (2007) Simultaneous detection and quantification of Ancylostoma duodenale, Necator americanus, and Oesophagostomum bifurcum in fecal samples using multiplex real-time PCR. Am J Trop Med Hyg 77(4): 685-690.
102.Xu J, Rong R, Zhang HQ, Shi CJ, Zhu XQ, Xia CM (2010) Sensitive and rapid detection of Schistosoma japonicum DNA by loop-mediated isothermal amplification (LAMP). Int J Parasitol 40(3): 327-331.
103.Yamamura M, Makimura K, Ota Y (2009) Evaluation of a new rapid molecular diagnostic system for Plasmodium falciparum combined with DNA filter paper, loop-mediated isothermal amplification, and melting curve analysis. Jpn J Infect Dis 62(1): 20-25.
104.Yamasaki H, Allan JC, Sato MO, Nakao M, Sako Y, Nakaya K, Qiu D, Mamuti W, Craig PS, Ito A (2004) DNA differential diagnosis of taeniasis and cysticercosis by multiplex PCR. J Clin Microbiol 42(2): 548-5453.
105.Yamasaki H, Nakao M, Sako Y, Nakaya K, Sato MO, Ito A (2006) Mitochondrial DNA diagnosis for taeniasis and cysticercosis. Parasitol Intl 55:S81-S85.
106.Zarlenga DS, Barry Chute M, Gasbarre LC, Boyd PC (2001a) A multiplex PCR assay for differentiating economically important gastrointestinal nematodes of cattle. Vet Parasitol 97(3): 199-209.
107.Zarlenga DS, Chute MB, Martin A, Kapel CM (1999) A multiplex PCR for unequivocal differentiation of all encapsulated and non-encapsulated genotypes of Trichinella. Int J Parasitol 29(11): 1859-1867.
108.Zarlenga DS, Chute MB, Martin A, Kapel CM (2001b) A single, multiplex PCR for differentiating all species of Trichinella. Parasite 8(2 Suppl):S24-6.
109.Zhang H, Thekisoe OM, Aboge GO, Kyan H, Yamagishi J, Inoue N, Nishikawa Y, Zakimi S, Xuan X (2009) Toxoplasma gondii: sensitive and rapid detection of infection by loop-mediated isothermal amplification (LAMP) method. Exp Parasitol 122(1): 47-50.
DIRECTORY OF MOLECULAR APPROACHES USING MULTIPLEX DNA SYNTHESIS FOR IDENTIFICATION AND DISCRIMINATION OF PARASITES
Le Thanh Hoa Insitute of Biotechnology SUMMARY Morphological examinations using phenotypic features for taxonomic identification are common techniques and huge achievements have been obtained in diagnosis and discrimination of parasites, but a number of facings are the difficulties of accurate discrimination of species/subspecies or their different lifecycle-developments (eggs, metacercariae…). During the past two decades, novel technologies have made molecular biology into diverse applications and new directories of development. The most distinguishing technologies of DNA amplification first to be mentioned are the PCR (polymerase chain reaction) and LAMP (loop-mediated isothermal amplification). These plural primer-based techniques (multiplex-PCR) and LAMP have been excellent tools to be globally applied in biomedical sciences, including molecular diagnosis of parasites. Both technologies, ie. PCR/RT-PCR (multiplex) and LAMP/RT-LAMP, are using: i) A number of primers (more than two); ii) DNA/RNA providing nucleic acid as template; iii) Deoxy-nucleotide triphosphate (dNTP) as structural materials; iv) Enzyme DNA polymerase to synthesize the DNA strands; v) Essential Magnesium ion (Mg++) as a member of components for the amplification reaction; vi) Visualization of DNA products for evaluation of the reaction in the right conditions. Each method has particular advantages in the accordingly conditions. In this review, we are addressing the importance of PCR and LAMP, principles of multiplex-PCR and LAMP and emphasis on the application of molecular approaches using multiplex DNA synthesis for identification and discrimination of parasites. This review is aimed to introduce up-dated investigations obtained over the world in recent 20 years based on generalization of literatures/databases in order to hope that the novel techniques/methods/technologies would be the good tools for the use in research and diagnosis of parasitic infections in Vietnam. Keywords: PCR, primer, multiplex-PCR, LAMP, linear/stem-loop DNA, parasites. ========================================================== PGS.TS Lê Thanh Hoà (Phòng Miễn dịch học, Viện Công nghệ Sinh học (18. Hoàng Quốc Việt, Cầu Giấy, Hà Nội, Việt Nam) ĐT: 04-37567297; 0912336855; Email: imibtvn@gmail.com) =====================================================
|

